上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
产品信息
EnGen SpRY Cas9 核酸酶来自化脓性链球菌(Streptococcus pyogenes),是一种经改造的、RNA 介导的 DNA 内切酶,可催化双链 DNA(dsDNA)的位点特异性切割。靶向需要约 100 个核苷酸的单链向导 RNA(sgRNA),与双链 DNA 底物上 PAM(Protospacer Adjacent Motif)上游的 20 nt 区域互补。EnGen SpRY Cas9 编码 11 个点突变(A61R、L1111R、D1135L、S1136W、G1218K、E1219Q、N1317R、A1322R、R1333P、R1335Q、T1337R),旨在降低对 PAM 的要求。与野生型 Spy Cas9 的经典 5´-NGG-3´ PAM 不同,SpRY Cas9 经证明在体外几乎没有 PAM 序列要求,能在许多具有 5´-NNN-3´ PAM 的位点上进行切割(尽管在体内与 5´-NYN-3´ PAM 相比,它表现出对 5´-NRN-3´ 的偏好)(1,2)。EnGen SpRY Cas9 核酸酶进行的 DNA 切割会在 PAM 上游 3 nt 处产生双链断裂。EnGen SpRY Cas9 核酸酶蛋白的 C 端含有猴病毒 40(SV40)T 抗原核定位序列(NLS)。
图 1:EnGen SpRY Cas9 核酸酶在体外对双链 DNA 靶向切割没有序列限制
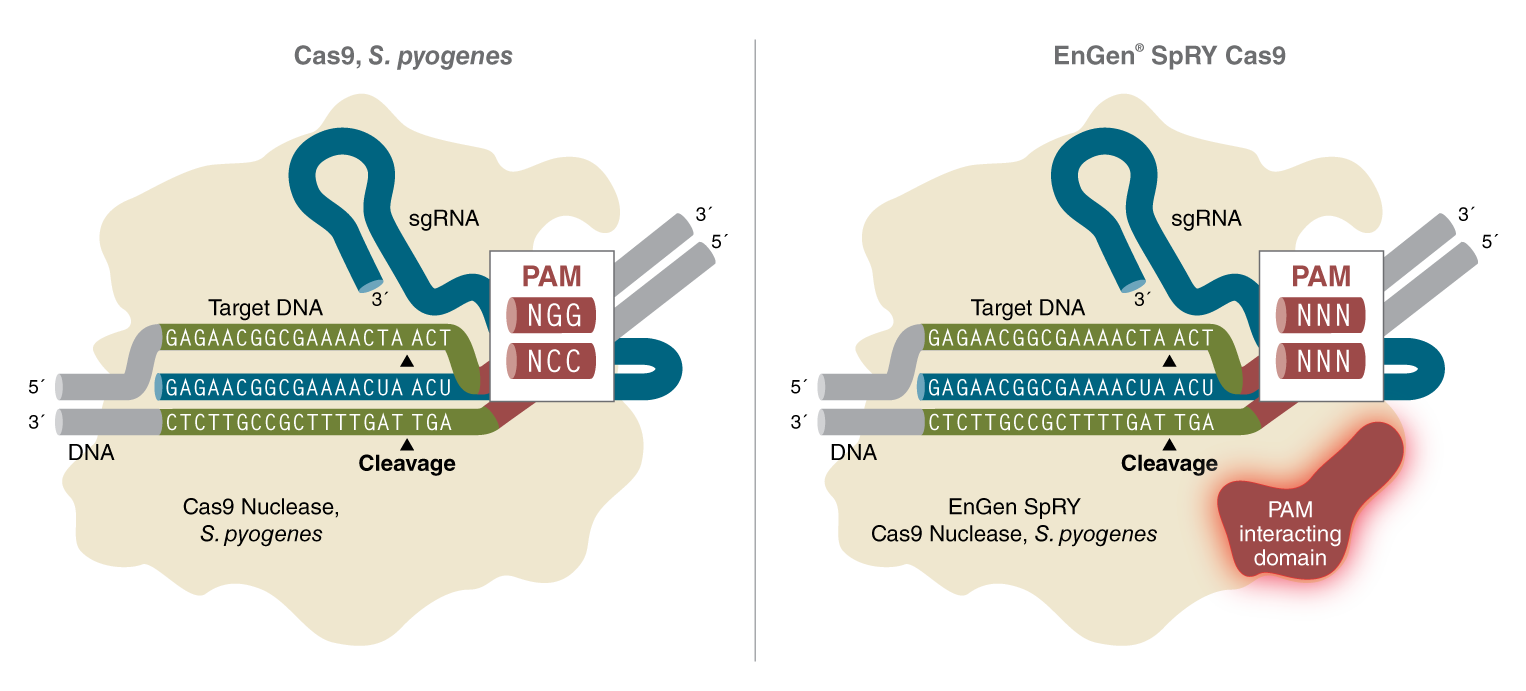
EnGen SpRY Cas9 是来自化酿脓链球菌的 Cas9 核酸酶的变体,在 PAM 作用结构域内有多个点突变(1)。与野生型 Cas9 不同,EnGen SpRY Cas9 不受 NGG PAM 的限制,在体外应用中,可以在任何三核苷酸序列上游 3 nt 处产生双链断裂。
图 2:使用 EnGen SpRY Cas9 核酸酶和 NEBuilder 高保真 DNA 组装预混液可以简化大载体克隆实验流程
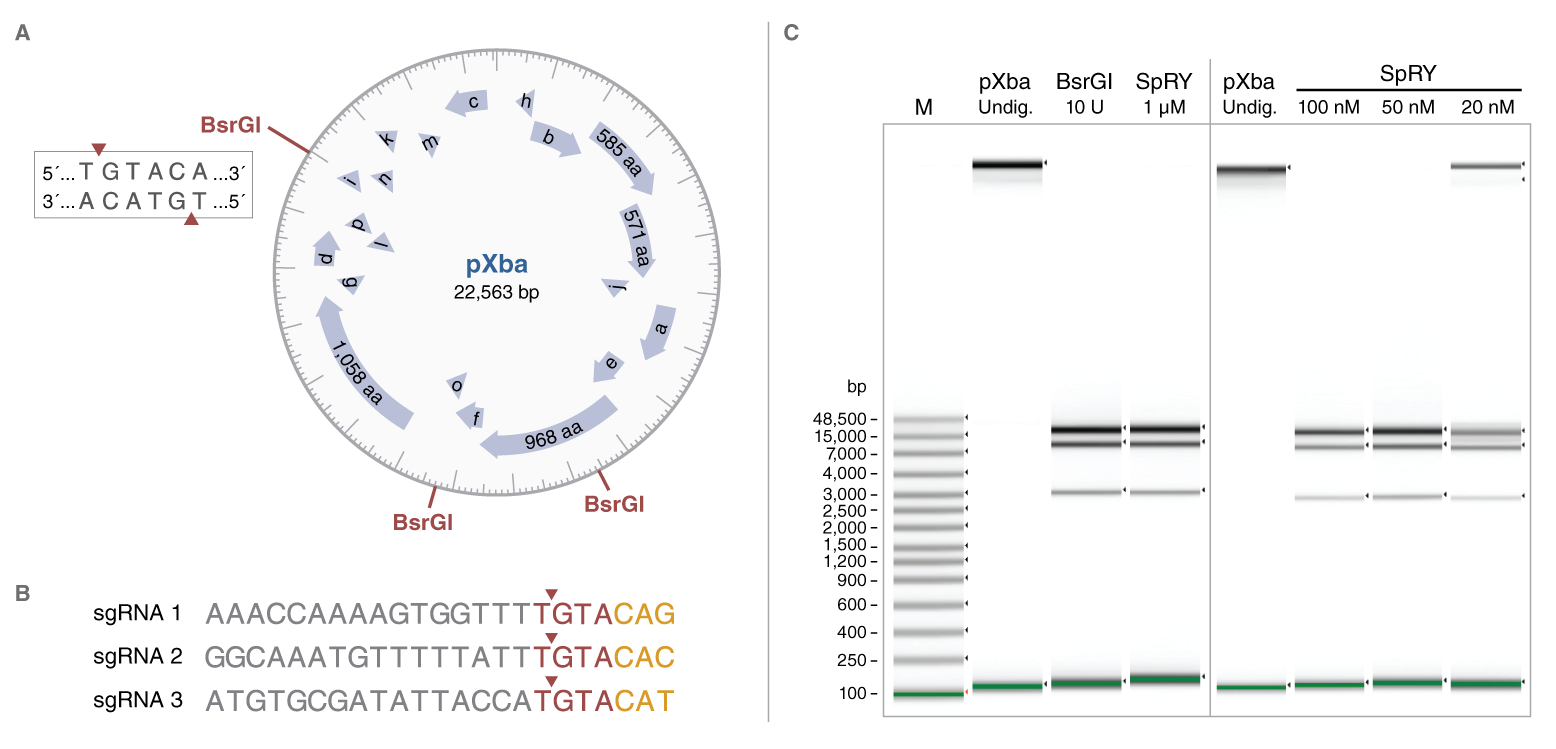 EnGen® SpRY Cas9 切割灵活性演示。A) 标有限制性内切酶 BsrGI 识别位点的 pXba 示意图。矩形框中显示了 BsrGI 识别序列,其切割位点用红色三角形表示。B) 用于靶向 pXba 中 BsrGI 位点的三种 sgRNA 序列。EnGen SpRY Cas9 和 BsrGI 的切割位点均用红色三角形表示。SpRY Cas9 非经典 PAM 以黄色文本表示。C) 在 1X NEBuffer r3.1 中,用 10 个单位的 BsrGI 或 1 µM EnGen SpRY Cas9 以及靶向 pXba 中所有三个 BsrGI 位点的三种 sgRNA 各 1 µM 消化 1 µg pXba(22,563 bp)。所有反应体系先在 37℃ 下温育 1 小时,然后再在 80℃ 下温育 5 分钟。使用 Agilent gDNA ScreenTape 系统,在 4200 TapeStation 仪器上通过凝胶电泳比较 BsrGI 和 SpRY 消化后产物 DNA 条带。BsrGI 和靶向 BsrGI 位点的 SpRY Cas9 对 pXba 的切割产生了几乎相同的条带图谱。使用浓度低至 50 nM 的 EnGen SpRY Cas9 和 sgRNA 消化 1 µg pXba 产生的切割与使用 1 µM 的结果相似。未消化的 pXba 作为对照进行电泳,但环状 DNA 在 gDNA ScreenTape 系统中不能精确的按照大小进行分离。垂直灰线标记了两个不同 ScreenTapes 的使用位置。
EnGen® SpRY Cas9 切割灵活性演示。A) 标有限制性内切酶 BsrGI 识别位点的 pXba 示意图。矩形框中显示了 BsrGI 识别序列,其切割位点用红色三角形表示。B) 用于靶向 pXba 中 BsrGI 位点的三种 sgRNA 序列。EnGen SpRY Cas9 和 BsrGI 的切割位点均用红色三角形表示。SpRY Cas9 非经典 PAM 以黄色文本表示。C) 在 1X NEBuffer r3.1 中,用 10 个单位的 BsrGI 或 1 µM EnGen SpRY Cas9 以及靶向 pXba 中所有三个 BsrGI 位点的三种 sgRNA 各 1 µM 消化 1 µg pXba(22,563 bp)。所有反应体系先在 37℃ 下温育 1 小时,然后再在 80℃ 下温育 5 分钟。使用 Agilent gDNA ScreenTape 系统,在 4200 TapeStation 仪器上通过凝胶电泳比较 BsrGI 和 SpRY 消化后产物 DNA 条带。BsrGI 和靶向 BsrGI 位点的 SpRY Cas9 对 pXba 的切割产生了几乎相同的条带图谱。使用浓度低至 50 nM 的 EnGen SpRY Cas9 和 sgRNA 消化 1 µg pXba 产生的切割与使用 1 µM 的结果相似。未消化的 pXba 作为对照进行电泳,但环状 DNA 在 gDNA ScreenTape 系统中不能精确的按照大小进行分离。垂直灰线标记了两个不同 ScreenTapes 的使用位置。
图 3:使用 EnGen SpRY Cas9 对 22.6 kb 质粒进行线性化,然后使用 NEBuilder 高保真 DNA 组装预混液进行克隆。
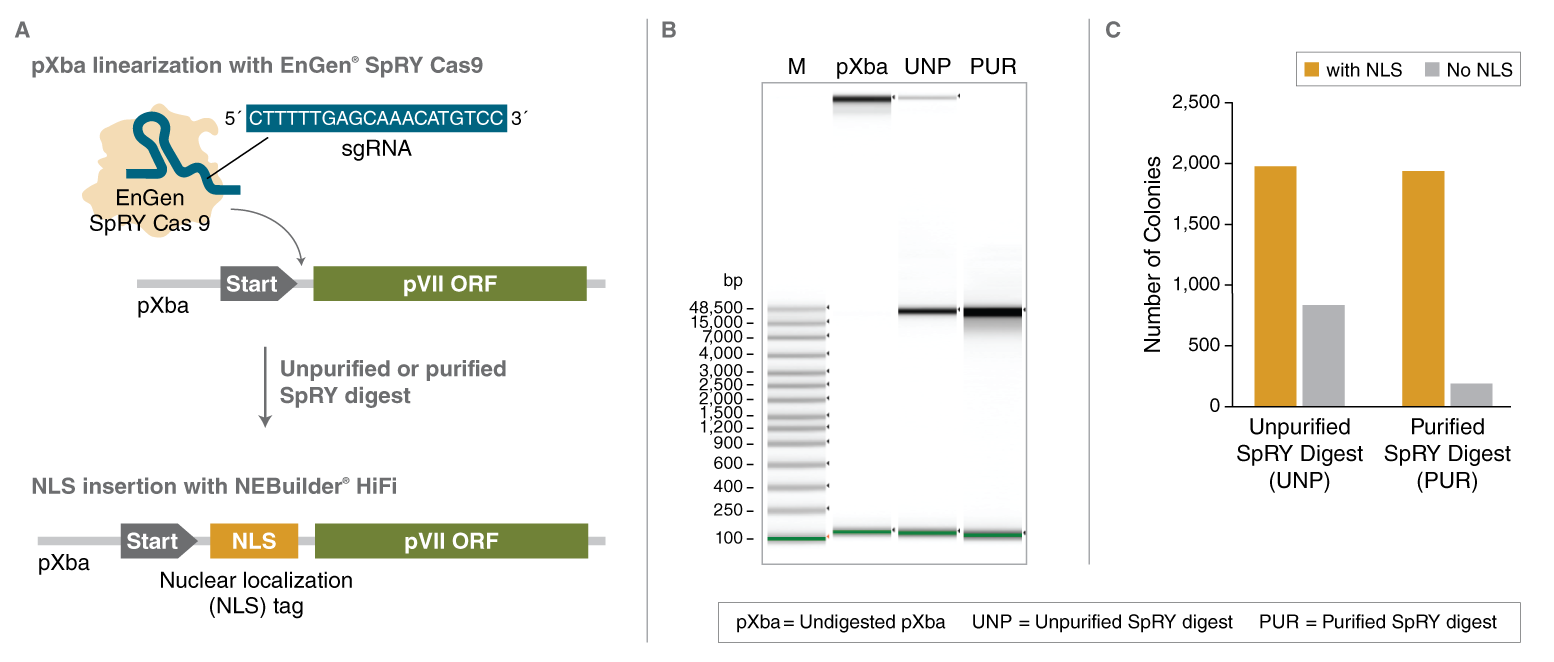 确定 EnGen SpRY Cas9 核酸酶消化产物是否适用于后续的 NEBuilder 高保真 DNA 组装预混液反应。A) 线性化和克隆方案的示意图。在 1X NEBuffer r3.1 中,使用 50 nM EnGen SpRY Cas9 和 50 nM sgRNA(靶向序列 5´-CTTTTTGAGCAAACATGTCC-3´),在 37℃ 条件下反应 1 小时,将 1 µg pXba(22,563 bp)在 pVII orf 起始密码子的下游进行线性化。在继续进行 DNA 组装之前,线性化质粒可以选择经过离心柱纯化或不纯化。根据推荐的操作说明,使用 NEBuilder 高保真 DNA 组装预混液试剂盒,将编码核定位序列(NLS)标签的寡核苷酸插入 pVII 中。B) 使用 Agilent gDNA ScreenTape 系统检查 EnGen SpRY Cas9 核酸酶消化后的线性化 pXba(纯化和未纯化的)。未消化的 pXba 作为对照进行电泳,但环状 DNA 在 gDNA ScreenTape 系统中不能精确的按照大小进行分离。C) 使用纯化或未纯化的 EnGen SpRY Cas9 消化产物进行 DNA 组装反应,统计转化后长出的克隆数。包括无插入片段对照,以定性评估 EnGen SpRY Cas9 未消化的质粒产生了多少转化子。在 DNA 组装反应之前,对线性化 pXba 的纯化降低了背景菌落的百分比。从纯化和未纯化 EnGen SpRY Cas9 消化产物中挑选 8 – 9 个转化子菌落,使用跨越插入片段的引物进行 PCR,以检查 DNA 组装是否正确。纯化和未纯化的 EnGen SpRY Cas9 消化产物,其产生的克隆 100% 显示正确的序列插入(数据在此未显示)。
确定 EnGen SpRY Cas9 核酸酶消化产物是否适用于后续的 NEBuilder 高保真 DNA 组装预混液反应。A) 线性化和克隆方案的示意图。在 1X NEBuffer r3.1 中,使用 50 nM EnGen SpRY Cas9 和 50 nM sgRNA(靶向序列 5´-CTTTTTGAGCAAACATGTCC-3´),在 37℃ 条件下反应 1 小时,将 1 µg pXba(22,563 bp)在 pVII orf 起始密码子的下游进行线性化。在继续进行 DNA 组装之前,线性化质粒可以选择经过离心柱纯化或不纯化。根据推荐的操作说明,使用 NEBuilder 高保真 DNA 组装预混液试剂盒,将编码核定位序列(NLS)标签的寡核苷酸插入 pVII 中。B) 使用 Agilent gDNA ScreenTape 系统检查 EnGen SpRY Cas9 核酸酶消化后的线性化 pXba(纯化和未纯化的)。未消化的 pXba 作为对照进行电泳,但环状 DNA 在 gDNA ScreenTape 系统中不能精确的按照大小进行分离。C) 使用纯化或未纯化的 EnGen SpRY Cas9 消化产物进行 DNA 组装反应,统计转化后长出的克隆数。包括无插入片段对照,以定性评估 EnGen SpRY Cas9 未消化的质粒产生了多少转化子。在 DNA 组装反应之前,对线性化 pXba 的纯化降低了背景菌落的百分比。从纯化和未纯化 EnGen SpRY Cas9 消化产物中挑选 8 – 9 个转化子菌落,使用跨越插入片段的引物进行 PCR,以检查 DNA 组装是否正确。纯化和未纯化的 EnGen SpRY Cas9 消化产物,其产生的克隆 100% 显示正确的序列插入(数据在此未显示)。
产品来源
大肠杆菌菌株,携带有克隆自化脓性链球菌(Streptococcus pyogenes)的 Cas9 基因,在其编码蛋白的 C 端含有猴病毒 40(SV40)T 抗原核定位序列(NLS),C 端同时带有 6xHis 标签
- 产品类别:
- Genome Editing Products,
- Programmable Nucleases Products
- 应用:
- Genome Editing Applications
-
产品组分信息
本产品提供以下试剂或组分:
| NEB # |
名称 |
组分货号 |
储存温度 |
数量 |
浓度 |
-
| |
EnGen® SpRY Cas9 |
M0669TVIAL |
-20 |
1 x 500 pmol |
20 µM |
| |
NEBuffer™ r3.1 |
B6003SVIAL |
-20 |
1 x 1.25 ml |
10 X |
|
-
| |
EnGen® SpRY Cas9 |
M0669MVIAL |
-20 |
1 x 2,500 pmol |
20 µM |
| |
NEBuffer™ r3.1 |
B6003SVIAL |
-20 |
1 x 1.25 ml |
10 X |
|
-
特性和用法
反应条件
1X NEBuffer™ r3.1
Incubate at 37°C
1X NEBuffer™ r3.1
100 mM NaCl
50 mM Tris-HCl
10 mM MgCl2
100 µg/ml Recombinant Albumin
(pH 7.9 @ 25°C)
贮存溶液
300 mM NaCl
10 mM Tris-HCl
0.1 mM EDTA
1 mM DTT
50% Glycerol
pH 7.4 @ 25°C
热失活
65°C for 5 minutes
-
注意事项
-
参考文献
- Walton, et al. (2020). Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science. Apr 17;368(6488), 290-6. DOI: 10.1126/science.aba8853
- Christie, et al. (2023). Precise DNA cleavage using CRISPR-SpRYgests. Nat. Biotechnol.. Mar 41, 409-16. DOI: 10.1038/s41587-022-01492-y
操作说明、说明书 & 用法
-
操作说明
- In vitro digestion of plasmid DNA with EnGen SpRY Cas9 (NEB #M0669)
FAQs & 问题解决指南
-
FAQs
- Why is it called EnGen SpRY Cas9?
- What is the difference between EnGen SpRY Cas9, EnGen Spy Cas9 HF1 (NEB #M0667) and EnGen Spy Cas9 NLS (NEB #M0646)?
- How do I design a single guide RNA for use with EnGen SpRY Cas9?
- How do I dilute the enzyme to 1 μM for in vitro reactions?
- Why do I observe incomplete digestion/editing?
- Why does digestion/editing efficiency differ between two different guide RNAs?
- Is it necessary to clean up my reaction before using the digestion products with NEBuilder?
- Where is the nuclear localization signal on EnGen SpRY Cas9 located?
- Which nuclear localization signal is fused to EnGen SpRY Cas9?
- Does NEB provide plasmids for guide RNA cloning?
- Can you tell me more about the switch from BSA to Recombinant Albumin (rAlbumin) in NEBuffers?



