上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
产品信息
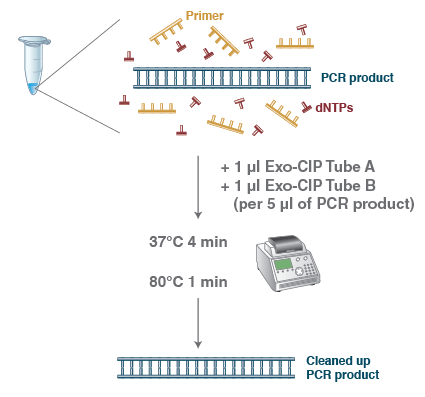
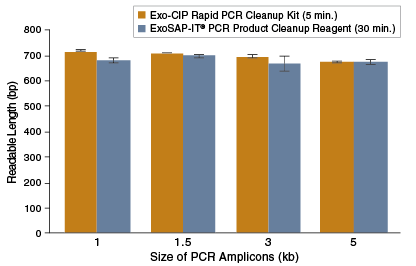
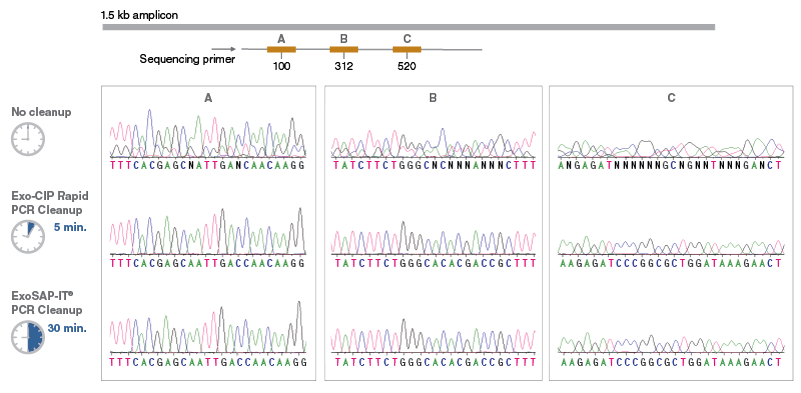
- 产品类别:
- DNA Cleanup Products,
- Nucleic Acid Purification Products
- 应用:
- PCR & Reaction Cleanup,
- DNA Amplification, PCR & qPCR
-
试剂盒组成
本产品提供以下试剂或组分:
NEB # 名称 组分货号 储存温度 数量 浓度 -
E1050S -20 Exo-CIP Tube A M1051AVIAL -20 1 x 0.1 ml 5,000 units/ml Exo-CIP Tube B M1052AVIAL -20 1 x 0.1 ml 1,250 units/ml
-
E1050L -20 Exo-CIP Tube A M1051AAVIAL -20 1 x 0.4 ml 5,000 units/ml Exo-CIP Tube B M1052AAVIAL -20 1 x 0.4 ml 1,250 units/ml
-
操作说明、说明书 & 用法
-
操作说明
- Protocol for Exo-CIP™ Rapid PCR Cleanup (#E1050)
- Enzymatic PCR Cleanup Protocol (NEB #M0525)
- Enzymatic PCR Cleanup Protocol (NEB #M0371)
-
应用实例
- Rapid Analysis of Genome Editing Efficiency using PCR Amplicons processed by the Exo-CIP™ Rapid PCR Cleanup Kit followed by Sanger sequencing
FAQs & 问题解决指南
-
FAQs
- Do I have to purify my PCR amplicon after cleanup?
- How should I store my treated samples after the enzymatic cleanup reaction?
- Why are my sequence reads less than 600 bases even when the electropherogram looks great?
- Why do I see two peaks (one large and one small) overlapping in one position of the electropherogram?
- Is my master mix / PCR buffer compatible with the Exo-CIP Rapid PCR Cleanup Kit?
