上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
产品信息
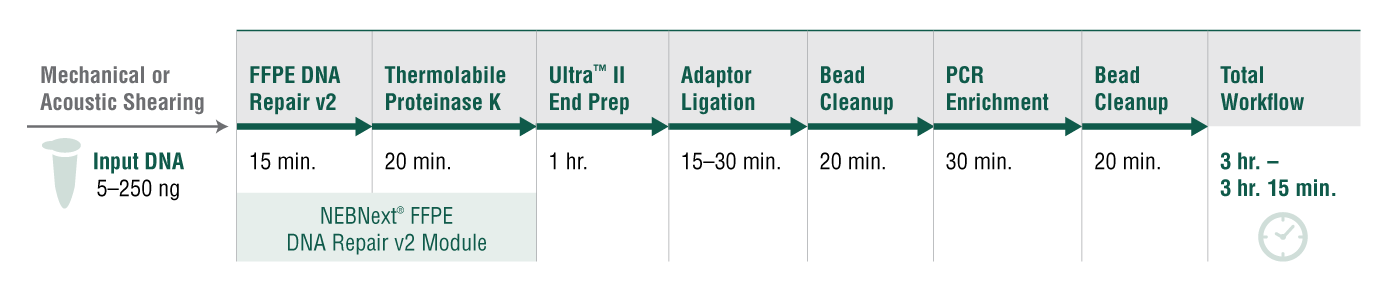
NEBNext FFPE DNA 修复模块 v2 包含经过优化的酶和缓冲液,可在二代测序流程中以精简的方式修复 FFPE DNA。
NEBNext FFPE DNA 修复模块 v2 在初代 NEBNext FFPE DNA 修复混合液基础上提升了性能:
- 更高的 FFPE DNA 修复效率
- 更精简的 NGS 文库制备流程
- 更便捷的反应缓冲液,包含高效 FFPE DNA 修复和下游末端修复以及 dA 加尾所需的所有缓冲液成分。
- 修复后使用热敏蛋白酶 Proteinase K 处理,无需纯化即可进行文库制备。
表 1:FFPE DNA 损伤类型以及 NEBNext FFPE DNA 修复模块 v2 的修复能力
| FFPE 损伤类型 | 可被 FFPE DNA 修复模块 v2 修复 |
|---|---|
| 胞嘧啶脱氨成尿嘧啶 | 是 |
| 切刻和缺口 | 是 |
| 氧化碱基 | 是 |
| 3´ 末端封闭 | 是 |
| DNA 片段化 | 否 |
| DNA-蛋白交联 | 否 |
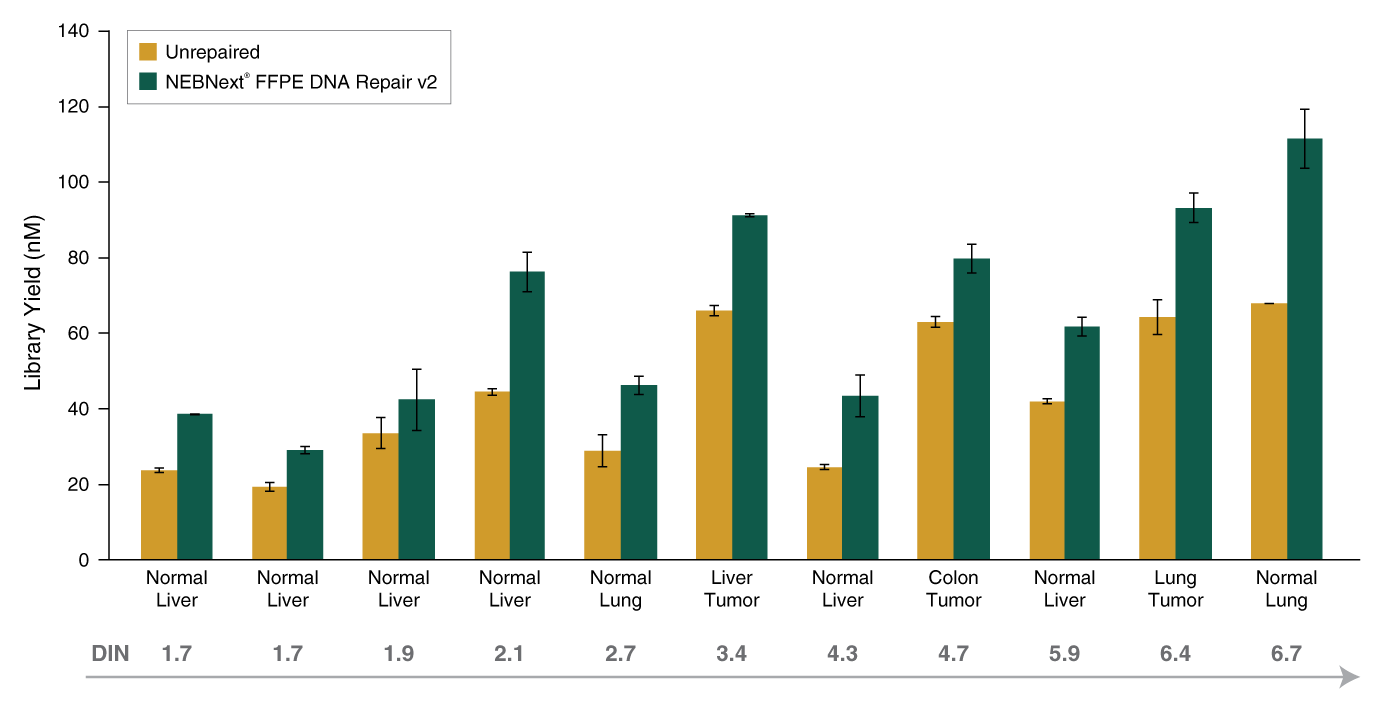


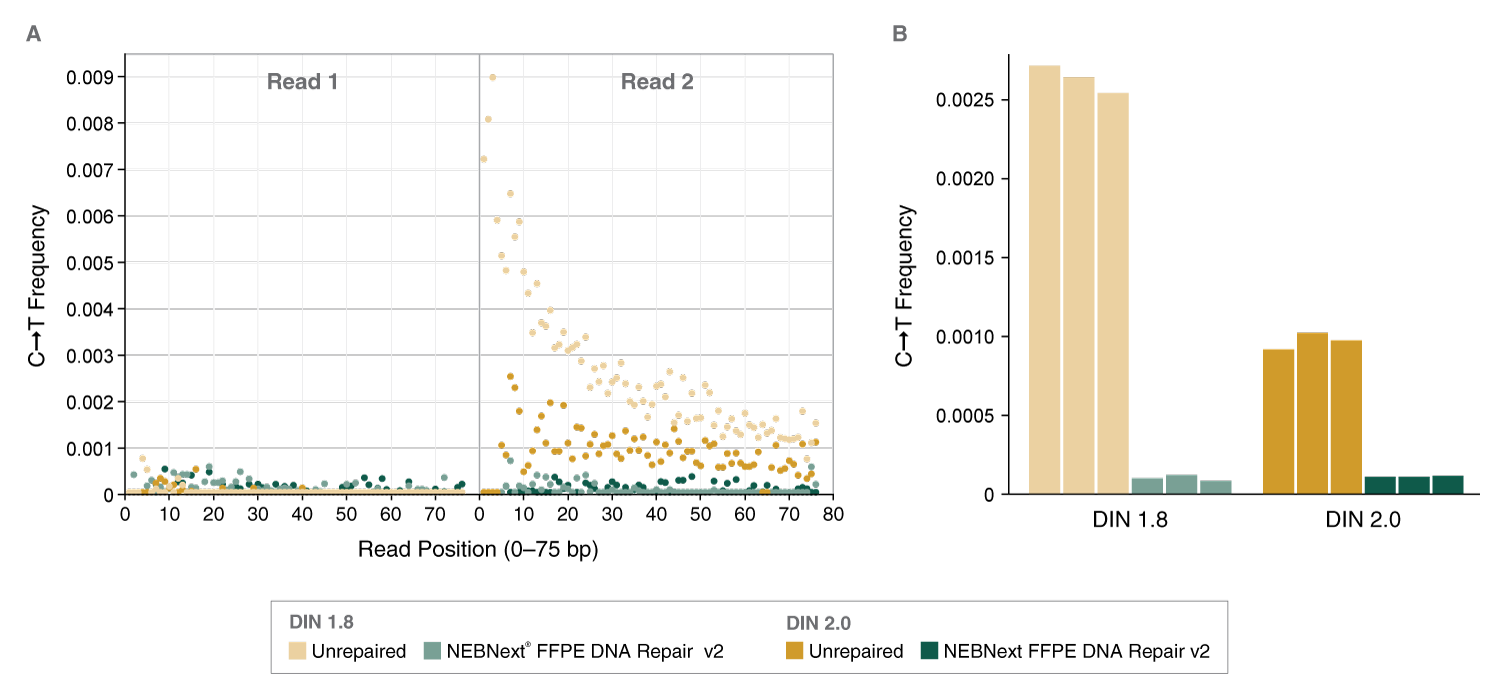
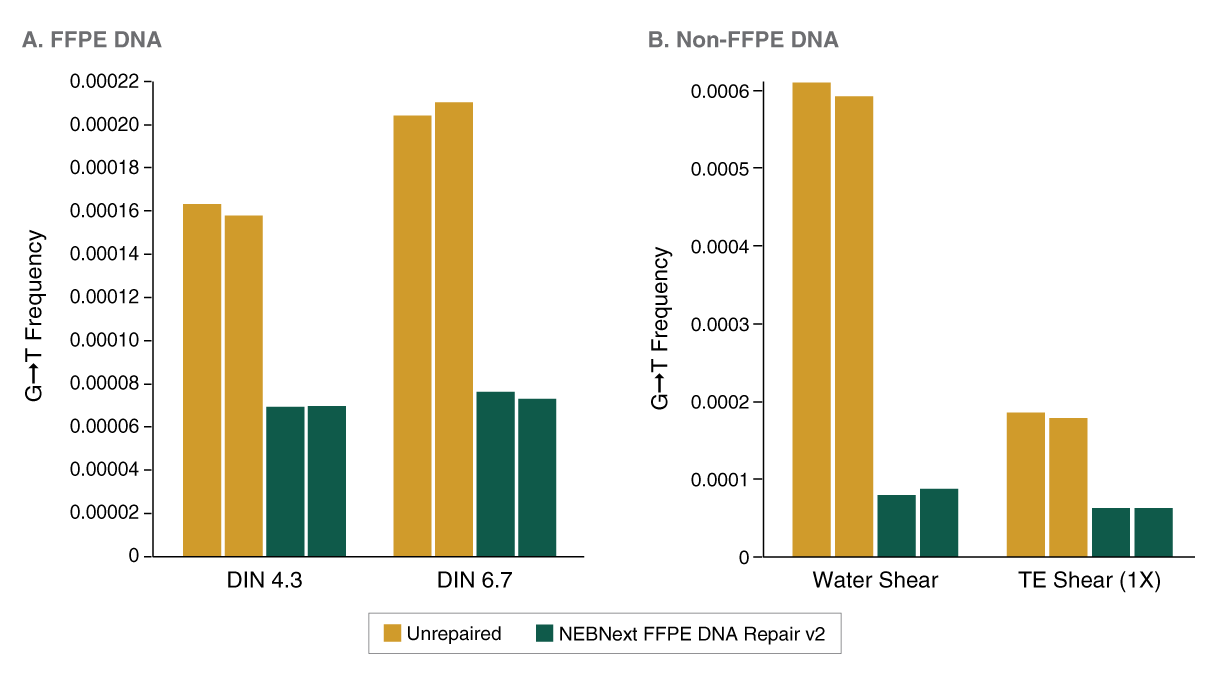
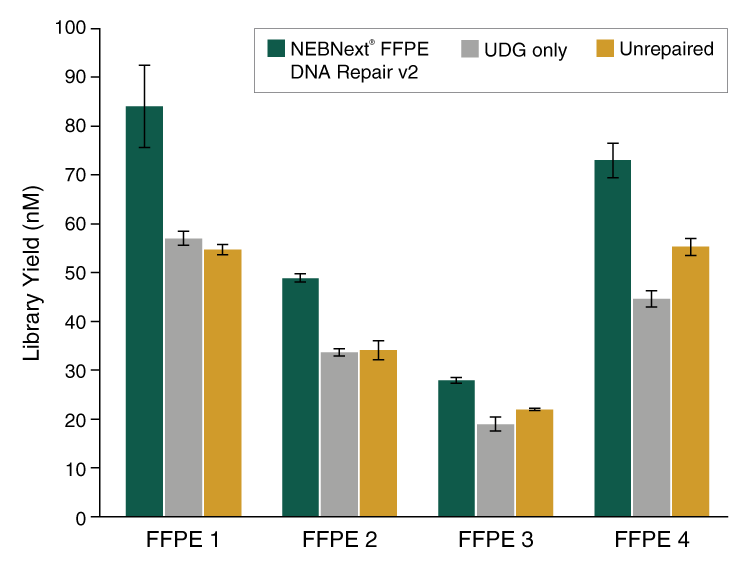
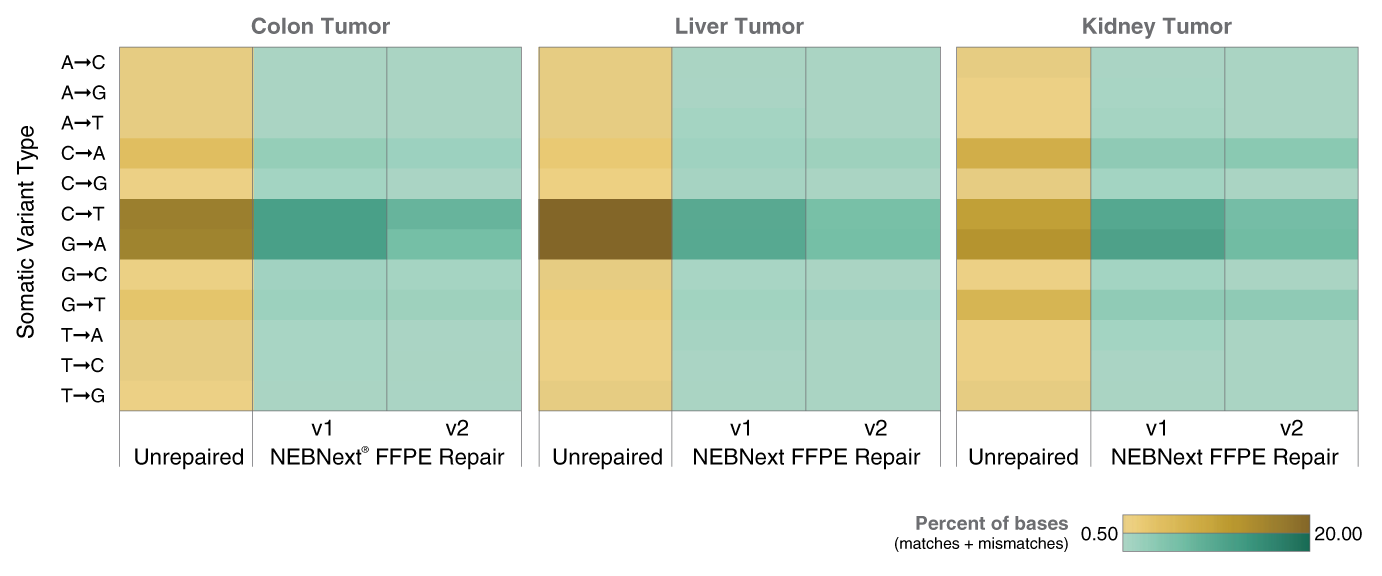
B:以在水* 或 pH 8.0 的 Tris-EDTA 中 Covaris 超声打断的 100 ng 人类基因组 DNA 为起始样本,使用 NEBNext FFPE DNA 修复模块 v2 和 NEBNext Ultra II DNA 文库制备试剂盒(NEB #E7645)制备了两个文库,每个样本重复 2 次。使用 Illumina NextSeq 500 对文库进行测序,向下抽样 200 万个 Reads,并按如上所述分析 GT 突变频率。*注意:厂商(Covaris)产品说明书不推荐在水中超声打断 DNA,但可用此方法生成含氧化损伤的底物。
- 产品类别:
- FFPE DNA Products,
- Next Generation Sequencing Library Preparation Products
-
试剂盒组成
本产品提供以下试剂或组分:
NEB # 名称 组分货号 储存温度 数量 浓度 -
E7360S -20 NEBNext® FFPE DNA Repair Mix v2 E7361AVIAL -20 1 x 0.048 ml Not Applicable NEBNext® Thermolabile Proteinase K E7362AVIAL -20 1 x 0.048 ml Not Applicable NEBNext® FFPE DNA Repair Buffer v2 E7363AVIAL -20 1 x 0.168 ml Not Applicable
-
E7360L -20 NEBNext® FFPE DNA Repair Mix v2 E7361AAVIAL -20 1 x 0.192 ml Not Applicable NEBNext® Thermolabile Proteinase K E7362AAVIAL -20 1 x 0.192 ml Not Applicable NEBNext® FFPE DNA Repair Buffer v2 E7363AAVIAL -20 1 x 0.672 ml Not Applicable
-
-
特性和用法
需要但不提供的材料
- NEBNext Ultra II DNA 文库制备试剂盒(NEB #E7645S、#E7645L、#E7103S 、或 #E7103L)或其它
- 80% 乙醇
- 无核酶水
- 0.1X TE(1 mM Tris-HCl pH 8.0,0.1 mM EDTA)
- 无 DNase、无 RNase PCR 联管
- DNA LoBind® 管(Eppendorf,#022431021)
- SPRISelect®试剂盒(Beckman Coulter 公司,#B23317)或 AMPure® XP Beads(Beckman Coulter 公司,#A63881)
- NEBNext 多样本接头引物试剂盒(www.neb.cn/oligos)
- 磁力架/支架(NEB #S1515,Alpaqua® #A001322,或同等产品)
- 热循环仪
- Agilent Bioanalyzer® 或 TapeStation®,以及相关试剂和耗材
- 接头稀释缓冲液 NEB #B1430S 或 NEBNext Unique 双端 Index 引物 UMI 接头稀释缓冲液,随 NEB #E7395S/L 提供
-
相关产品
相关产品
- NEBNext® Ultra™ II DNA 文库制备试剂盒
- NEBNext® Ultra™ II DNA 文库制备试剂盒(含纯化磁珠)
操作说明、说明书 & 用法
-
操作说明
- Where can I find guidelines and protocols for using the NEBNext FFPE DNA Repair v2 Module (NEB #E7360)?
-
说明书
产品说明书包含产品使用的详细信息、产品配方和质控分析。- manualE7360
工具 & 资源
-
Web 工具
- NEBNext Selector
FAQs & 问题解决指南
-
FAQs
- What is the difference between the NEBNext FFPE DNA Repair v2 Module (NEB #E7360) and the NEBNext FFPE DNA Repair Mix (NEB #M6630)?
- Will the FFPE DNA Repair v2 Module ligate my DNA fragments?
- Will treating my DNA with the FFPE DNA Repair v2 Module hurt my downstream reaction?
- Will the FFPE DNA Repair v2 Module blunt the ends of the DNA?
- Does the FFPE DNA Repair v2 Module insert random nucleotides into the sequence that it repairs?
- Does the FFPE DNA Repair v2 Module remove covalent modifications from DNA bases, such as biotin or digoxigenin?
- Does the FFPE DNA Repair v2 Mix repair DNA-protein crosslinks?
- Does the FFPE DNA Repair v2 Mix fix blocked 3′ ends?
- Can the FFPE DNA Repair v2 Mix repair damage in both single- and double-stranded DNA? Or, does it require double stranded DNA as a template?
- If I had a DNA template with mutation sites (i.e. 8-oxoguanine or deaminated cytosines) that are directly adjacent to each other on opposite strands would treatment with the FFPE DNA Repair v2 Mix cause a double strand nick/break?
- What gap lengths can be repaired with the FFPE DNA Repair v2 Mix?
- Can the FFPE DNA Repair v2 Module repair bisulfite-treated DNA?
- When working with fragmented DNA, will the ends be ligated together by the FFPE DNA Repair v2 Module?
- How are abasic sites repaired by the FFPE DNA Repair v2 Module?
- Can I use the FFPE DNA Repair v2 Module to repair other types of low quality DNA other than FFPE DNA?
